BPS Bioscience Inc.品牌商
5 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0.20000000000000018
- 0.20000000000000018
- 2.2
- 2.2
推荐产品
公司新闻/正文
PARP 蛋白和其检测方法-BPS Bioscience
7821 人阅读发布时间:2023-09-27 17:12
1.PARP 介绍
维持基因组完整性对于正常细胞功能至关重要。在人类中,超过 150 种蛋白质形成了一个复杂的 DNA 损伤反应 (DDR) 网络,该网络不断发现和修复 损伤的 DNA (1)。
其中,PARP (Poly ADPRibose Polymerase) 蛋白家族由 17 个成员组成,它们催化蛋白质的 ADP-核糖基化。PARP 涉及广泛的生物学功能,包括修复 DNA 损伤、维持基因组稳定性、调节染色质重塑、有丝分裂纺锤体组装、RNA 周转、基因表达调节以及 DNA 甲基化等 (2)。
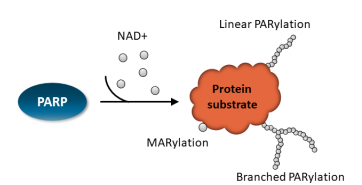
图1:单个和多个 ADP 核糖化
尽管这些蛋白质都属于同一家族,但它们显示出截然不同的特征。少数 PARP 仅具有单核糖基化活性(MARylation),而其他 PARP 催化以线性或分支模式发生的多核糖基化(PARylation)(图 1)。PARP 可能主要定位于细胞核、细胞质或两者,并且在大小和结构上存在显著差异,并包含多种多样的功能域(图 2)。值得注意的是,PARP5A 和 PARP5B 除了催化结构域外只有一个大的锚蛋白结构域(因此命名为端锚聚合酶 TNKS1 和 TNKS2,分别对应于 PARP5A 和 B)。其他显著特征包括 PARP1、PARP2 和 PARP3 的严格 DNA 依赖性,以及每种酶的底物特异性。
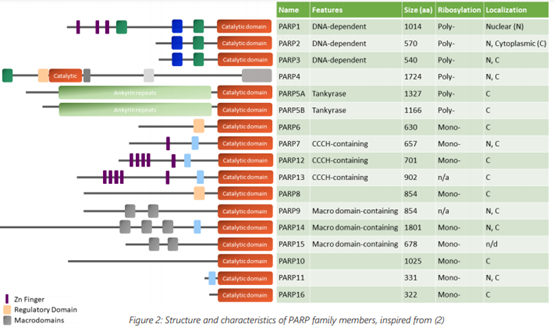
图2:PARP 家族成员的结构和特点,参考文献(2)
正如许多具有重要作用的蛋白质家族的情况一样,PARP 蛋白质在功能上有重叠。PARP1 和 PARP2 主要参与 DNA 修复,这两种蛋白都调节 DDR 网络。PARP2 还调节表观遗传、增殖和炎症过程,对精原细胞、胸腺和脂肪组织的发育很重要 (3, 4)。
相反,当 DNA 受损无法修复时,PARP1 会改变转录并诱导细胞凋亡。它是受损 DNA 的第一反应者,其重要性体现在其丰度上,因为它是最常见的核蛋白之一。
DDR通路中的缺陷会导致基因组不稳定,进而支持肿瘤细胞的出现和进化,因为这些缺陷会导致突变积累。因此,当DNA损伤修复和肿瘤抑制因子BRCA1或BRCA2(乳腺癌1/2型易感蛋白)发生突变时,会削弱细胞通过同源重组(HR)修复双链DNA断裂的能力,从而增加患上乳腺癌、卵巢癌或前列腺癌的风险(5)。
然而,丧失HR依赖性DNA修复系统意味着这些肿瘤细胞依赖于其他修复途径来生存,从而暴露它们的治疗致命弱点。实际上,人们对PARP作为治疗靶点的兴趣最初源于发现抑制PARP1/2可以杀死具有BRCA1或 BRCA2 突变的癌细胞。这一观察首次证明了合成致死率的概念,即由于两种蛋白质同时破坏而导致的细胞死亡,这两种蛋白质在单独受损时不会导致活力丧失。
目前已有几种 PARP 抑制剂被批准用于临床,许多其他抑制剂也正在通过(前)临床阶段(3)。由于阻断肿瘤细胞中的任何HR通路(不限于BRCA基因)都会产生"BRCAness"(6),因此这种治疗应用也在不断扩大。然而,改进现有抑制剂、靶向其他 PARP 家族成员并添加新的抑制剂以规避治疗耐药性仍然是癌症药物开发的重点(7)。
使用 BioRender.com 创建

图3:PARP 介导的蛋白底物的 ADP 核糖基化
ADP-核糖基化是利用NAD+作为核糖供体,在蛋白质底物中的 Glu、Asp 或 Lys 残基的羧基上可逆地添加 ADP 核糖单元(如图3所示)。为了测量 PARP 活性,需要使用 PARP 底物、NAD+、DNA 依赖性 PARP 1-3的 DNA 探针和纯化的 PARP 酶。为确保检测的灵敏度、稳健性和可重复性,必须仔细优化这些组件。以下几点需要注意:
- l 蛋白质必须具有酶促活性和纯化,没有会改变其活性的污染物。构建带有标签的重组蛋白有利于亲和纯化。
- l 对于每个新批次的蛋白质都应进行蛋白质酶活性测试,以确保随着时间的推移测定的一致性。
- l 在测定开发阶段,需要进行蛋白质滴定,以确定每个 PARP 测定中使用的最佳浓度。
- l 确定 PARP1、PARP2 和 PARP3 的最佳 DNA 探针可提高检测特异性。
- l 在基于标记的 NAD+ 检测中,确定合适的 NAD+ 混合物对于检测的灵敏度至关重要。这必须为每种酶确定,以解释单核糖基化或多核糖基化和酶动力学。

在设计用于筛选大型化合物库的测定时,通量、步骤数和低体积是关键标准。但是,对于小型研究实验室进行分析,总体分析成本或仪器可用性可能是最重要的方面。在选择测定方法时时,需要考虑以下因素:
• 仪器可用性
• 使用方便程度
• 成本
• 灵敏度
• 通量
• 完成所需时间
化学发光和比色测定试剂盒是基于酶联免疫吸附测定(ELISA)的,旨在测量药物分析应用中的 PARP 活性。在这些测定中,将底物蛋白包被在板上(见图4),然后将生物素化的 NAD+混合物与纯化的 PARP酶一起添加到优化的测定缓冲液中。链霉亲和素 -HRP(辣根过氧化物酶)处理板后,添加适当的HRP底物以产生化学发光或颜色,然后在每个步骤后清洗板。信号强度与附着在组蛋白上的生物素-NAD+的数量成正比。
考虑到 PARP1 和 PARP2 之间的高度同源性,很难找到对PARP1具有比对PARP2更好的亲和力的药物。这是理想的,因为 PARP2 的抑制会导致更严重的副作用(8)。为了减少脱靶活性,研究人员正在筛选靶向PARP1且亲和力高于 PARP2 的分子。在一组比较 AZD5305 和 Olaparib 功效的实验中,BPS Bioscience 的科学家能够显示出对PARP1和PARP2的独特抑制作用。实际上,这两种抑制剂对 PARP1 显示出相似的 IC50(7和8 nM)。在分析 PARP2 时,Olaparib 的 IC50 为 0.3 nM,而 AZD5305 的 IC50为100 nM,证明了该分析的精确特异性和灵敏度。

图4:PARP1-3 和 TNKS1-2 催化多核糖基化的化学发光 ELISA 的原理(上部分)。使用 PARP1 和 PARP2 化 学发光 ELISA 试剂盒 BPS Bioscience#80551 和 #80552 的代表性结果(下部分)。
AlphaLISA® 是一种基于微珠的免洗技术,由 PerkinElmer 开发,可用于定量蛋白质-蛋白质结合或新酶产物的测量。 AlphaLISA® PARP 均相检测试剂盒采用高度特异性的 PAR 化底物抗体来识别 PARP 家族成员的酶活性。测定的特异性取决于纯化蛋白的鉴定。
测定方案非常简单:首先将酶与生物素化底物孵育。然后添加受体珠和一抗,最后添加供体珠。这些免洗步骤之后直接读取 Alpha 计数即可。需要注意的是,这些检测需要使用专门的 AlphaScreen 酶标仪。这种测定设计因其快速完成、高效和适合高通量应用而备受青睐,例如筛选小分子文库以识别新的 PARP 抑制剂。此外,它还可以用于准确测量化合物的 EC50。
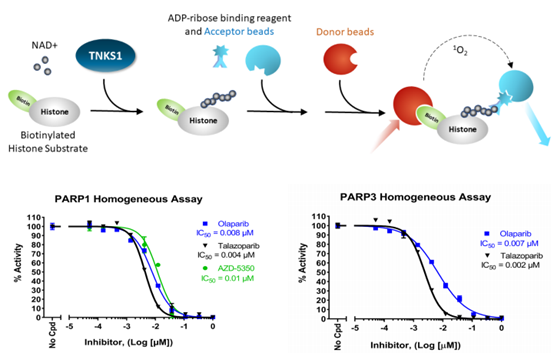
图5:AlphaLISA® PARP均相测定的原理(上部分)。通过使用Homogeneous Assay Kits BPS Bioscience #78438和#78491(下部分),通过增加所示抑制剂的浓度测量PARP1或PARP3活性的抑制。
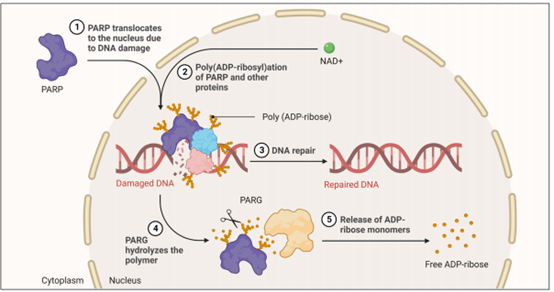
当受损的 DNA 结合 PARP1和 PARP2 时,它们会将 PAR 链添加到它们自己的蛋白质主链上(autoPARylation),然后再添加到其他 DDR 蛋白质上,以招募和激活它们(8)。PARylated的PARP1 PARP随后从 DNA 上分离,以便其他 PARylated 的伙伴可以启动修复过程。目前已批准的一些药物通过与 NAD+ 竞争结合催化位点,降低 PARP1 和 PARP2 的活性。如果没有 NAD+,PARP 就无法进行 PARylation 并保持与受损 DNA 的结合,使其免受其他 DDR 蛋白的影响。这会阻止 DNA 修复并增加细胞毒性,从而增强这些药物的作用。因此,这类药物的细胞毒性作用主要取决于它们将蛋白质捕获到受损 DNA 上的效率(9),尽管科学家最近发现,使用PARP抑制剂将 PARP1 (而不是PARP2)捕获到DNA会导致细胞毒性增加。因此,在筛选这些药物时,应包括量化PARP捕获能力和区分抑制剂对 PARP1 或 PARP2 的选择性的测定。
大多数商业上可用的 PARP 活性测定法量化目标蛋白质(例如组蛋白)的 PARylation,并且一次仅测试一种 PARP 酶。相比之下,PARPtrap™ 组合检测试剂盒用于 PARP1 和 PARP2,可以比较同一检测中分子捕获 PARP1 和 PARP2 的能力。
该测定使用荧光标记的 DNA 探针,根据 PARP1 或 PARP2 结合发射偏振光。当 PARP1 或 PARP2 与 DNA 结合时,这些探针具有高荧光偏振(FP)。当科学家将 NAD+ 添加到检测中时,PAR 化酶从探针上分离,从而降低 FP水平。相反,如果他们添加 NAD+ 和 PARP 抑制剂,则抑制剂的捕获能力会以剂量依赖的方式增加 FP。

这种同质、简单的测定法可以结合到高通量药物发现筛选中,以筛选可增强 DNA 上 PARP1 或 PARP2 捕获的分子。PARPtrap™ 检测让研究人员能够高效地筛选他们的文库,寻找最特异和最有效的抑制剂。

图6:代表性结果展示了药物诱导的PARP1和PARP2捕获(BPS Bioscience #80584和#78296)。这种创新性的测定旨在筛选小分子PARP抑制剂,这些抑制剂可以在DNA上捕获酶,这是PARP1/2独有的特征。
靶向嵌合体或PROTAC的蛋白水解是一种新兴的蛋白质降解技术,它可以通过将感兴趣的蛋白质靶向泛素E3连接酶来促进蛋白酶体介导的蛋白质降解。与传统的小分子介导的蛋白质活性抑制相比,这项新技术具有明显的优势。
PROTAC分子由结合E3连接酶的配体组成,通过接头连接到结合目标蛋白质的配体。一旦形成这样的复合物,它将被引导到蛋白酶体,从而促进目标蛋白质的降解。与传统的药物相比,单个PROTAC可以促进许多蛋白质的降解,因为它会在其目标降解后被回收。
PROTAC的优点在于可以对“难以药物化”的蛋白质进行靶向,这使得它成为一种潜在的替代策略。任何感兴趣的蛋白质都可以使用这种技术作为目标。但是,PROTAC的开发需要几个优化步骤,这些步骤因量化蛋白质降解的技术难度而减慢。
PROTAC优化分析通过直接测量PROTAC介导的复合物形成来绕过这些困难。这种方法能够提供更准确和可靠的结果,从而加速PROTAC的开发和优化过程。

测定原理:所测的PROTAC(这里是阳性对照iRucaparib-AP6)与PARP1和CRBN相互作用,使它们靠近。PARP1含有一个被GSH供体珠识别的GST标记,而CRBN含有一个与抗FLAG抗体结合的FLAG标记的接受体珠。在激发供体珠后,供体珠会产生单线态氧。单线态氧会激发受体珠,并根据相互作用水平发射光。

图7,左侧图:在固定PARP1浓度下,在存在或不存在固定iRucaparib-AP6(PROTAC)BPS Bioscience #78441的条件下滴定cereblon。右侧图:通过增加Rucaparib(PARP1抑制剂)或Pomalidomide(CRBN抑制剂)的浓度,抑制iRucaparib-AP6介导的Cereblon与PARP1的相互作用。
BPS Bioscience提供灵敏、稳健的检测,可以在短时间内提供高质量的数据,以满足您的研究需求。我们支持比较药物对所有 PARP 家族成员的 IC50 和筛选 PARP 捕获药物等研究。我们会及时提供协议、原始数据和分析数据,以确保您获得高质量的数据。我们的检测广泛涵盖 PARP 酶组合,有助于进行整个 PARP 家族的化合物库筛选、新检测开发和优化以及 IC50 测定等研究。除此之外,我们还提供独特的 PARPtrapTM 分析,可专门评估药物将 PARP1 或 PARP2 捕获到 DNA 的效力。无论您的研究需求是什么,我们都能够为您提供最优质的服务。
应用实例
Wang H. 等人发表的研究论文介绍了一种处于临床开发阶段的强效选择性 PARP1/2 抑制剂 Pamiparib (BGB-290)。该药物已在临床前模型中证明具有 PARP-DNA 捕获能力以及强大的抗肿瘤活性和 CNS 渗透能力。
本研究使用了每种化学发光检测试剂盒的相应检测缓冲液和溶液,针对单个 PARP 蛋白滴定 Pamiparib,包括 PARP1/2/3、PARP5A 和 PARP5B。此外,BPS Bioscience 还对 PARP6、PARP7、PARP8、PARP10、PARP11 和 PARP12 进行了测试。
结果显示,Pamiparib 对 PARP1 和 PARP2 的选择性相等,EC50 分别为 1.3 nM 和 0.92 nM,对 PARP3 的效力大约低 50 倍,并且对其他 PARP 家族成员表现出非常低的亲和力。因此,Pamiparib 是一种有效的选择性 PARP1 和 PARP2 抑制剂。

为了方便使用,我们提供了测定试剂盒成分的单独购买,包括底物和缓冲液。我们提供了约30种PARP抑制剂,可用作对照或优化检测。
此外,我们提供了一套包含八种PARP抑制剂的样品,包括广泛结合特异性的抑制剂,例如奥拉帕尼、尼拉帕尼、Rucaparib、他拉唑帕尼、维利帕尼。其中,AZD5305对PARP1具有特异性,XAV939对PARP5A和PARP5B具有特异性,而RBN-2397对PARP7具有特异性(PARP抑制剂组,BPS Bioscience #78318)。
BPS Bioscience 提供PARP家族成员最多的选择。我们提供纯化的、带标签的重组蛋白,这些蛋白具有酶促活性,适用于分析开发和抑制剂筛选或分析(例如,IC50 测定)。
我们还提供众多的 PARP 检测试剂盒和服务系列,包括独特的 PARPtrap 检测,有助于评估药物的特异性和有效性,并可用于研究其作用机制。
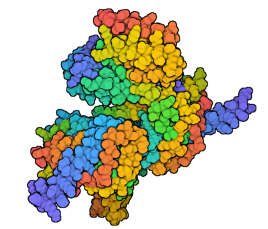
人类PARP1结构域(Zn1、Zn3、WGR、HD)与DNA双链断裂结合的结构。PDB ID:7s81,Rouleau-Turcotte等人,Mol Cell(2022),PMID:35793673。使用BioRender.com创建。
参考文献:
1. Wood RD, et al. Human DNA repair genes. Science, 291: 1284-1289 (2001); PMID: 15922366.
2. Ummarino S, Hausman C, Di Ruscio A. The PARP Way to Epigenetic Changes. Genes, 12(3): 446 (2021); PMID: 33804735.
3. Rose M, et al. PARP inhibitors: clinical relevance, mechanisms of action and tumor resistance. Front. Cell Dev. Biol., 8: 564601 (2020); PMID: 33015058.
4. Ali SO, et al. Understanding specific functions of PARP2: new lessons for cancer therapy. Am. J. Cancer Res., 6(9): 1842-1863 (2016); PMID: 27725894.
5. Lord CJ, Ashworth A. BRCAness revisited. Nat. Rev. Cancer, 16(2): 110-120 (2016); PMID: 26775620.
6. Hu Y, Guo M. Synthetic lethality strategies: Beyond BRCA1/2 mutations in pancreatic cancer. Cancer Sci., 111(9): 3111-3121 (2020); PMID: 32639661.
7. Rudolph J, et al. Inhibitors of PARP: Number crunching and structure gazing. PNAS USA, 119(11): e2121979119 (2022); PMID: 35259019.
8. Slade D, Eustermann S. Tuning drug binding. Science, 368(6486): 30-31 (2020); PMID: 32241937.
9. Pommier Y, et al. Laying a trap to kill cancer cells: PARP inhibitors and their mechanisms of action. Sci. Transl. Med., 8(368): 368er7 (2016); PMID: 27797957.
BPS Bioscience Inc.提供相关的产品和检测服务。



